- Trasparenza
- Amministrazione trasparente
- Concorsi e collaborazioni
- Frequenza Volontaria e Tirocini
- Appalti
- Regolamenti
- Ufficio Relazioni con il Pubblico
- Protocollo Informatico
- Albo Aziendale Informatico
- Albo Aziendale Informatico (storico)
- Albo Esperti
- Albo Fornitori
- Albo Professionisti
- Segnalazione e/o reclami U.R.P.
- Segnalazione illeciti (whistleblowing)
- Privacy e protezione dei dati personali
- Normativa di riferimento dell'Istituto
- Comitato Unico di Garanzia
- 5 per mille IZSAM
- Cookie Setting
- Ambiente
- Biologia delle acque
- Centro Ricerche per gli Ecosistemi marini e Pesca
- Termoli Sea Cleaners
- Conferenza stampa di presentazione
- Entra nel vivo il progetto Termoli Sea Cleaners
- Termoli Sea Cleaners all'Università del Molise
- Termoli Sea Cleaners spiegato agli alunni della III elementare
- Termoli Sea Cleaners. Al via il primo incontro divulgativo per presentare le attività del progetto
- Raccolta rifiuti marini Termoli Sea Cleaners
- Sea cleaners: prosegue il progetto "Spazzini del mare" - video
- Eccellenza
- Regionali
- Centri nazionali
- Centri internazionali
- Centri di Collaborazione WOAH
- Laboratorio Referenza WOAH Brucellosi
- Laboratorio Referenza WOAH Pleuropolmonite Contagiosa Bovina
- Laboratorio Referenza WOAH Bluetongue
- Laboratorio Referenza WOAH West Nile Fever
- Centro Referenza FAO Epidemiologia Veterinaria
- Centro Referenza FAO Coronavirus Zoonotici
- Centro Referenza FAO Salute Animale e Sicurezza Alimentare
- Centro Referenza UE Benessere dei ruminanti e degli equidi
- Laboratorio Riferimento UE (EURL) per la Rift Valley Fever
- Laboratorio trasportabile
SARS-COV-2: PIÙ EFFICIENZA NELLA CACCIA ALLE VARIANTI
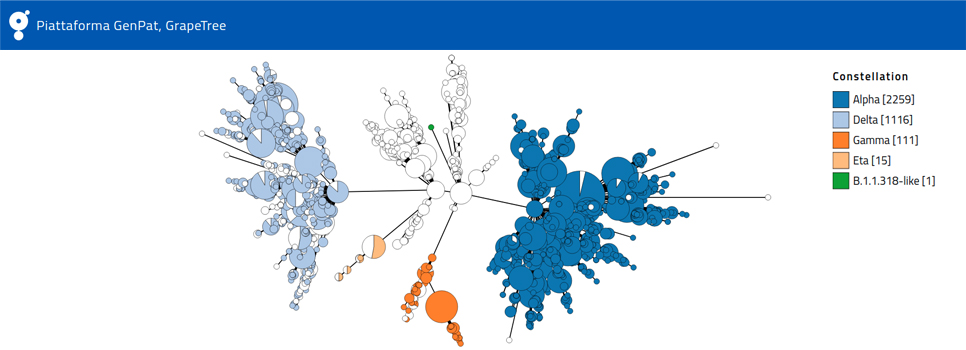
Un nuovo metodo di analisi matematica permette di tipizzare i campioni con alta precisione e maggiore velocità. Un contributo importante al tracciamento del virus e della sua evoluzione
Nella lotta alla pandemia una delle principali priorità è seguire l’evoluzione del virus, individuando la comparsa di nuove varianti e valutandone la diffusione. Precisione e velocità delle analisi genomiche sono elementi fondamentali di questa sorveglianza, gli stessi al centro di una recente ricerca dell’Istituto Zooprofilattico Sperimentale dell’Abruzzo e del Molise.
I ricercatori del “ Centro di Referenza Nazionale per Sequenze Genomiche di microrganismi patogeni: banca dati e analisi di bioinformatica ” (GENPAT) dell’IZSAM hanno sviluppato un nuovo metodo matematico per analizzare e confrontare il genoma di grandi quantità di campioni virali. Pubblicato sulla rivista scientifica BMC Genomics, il metodo proposto permette di identificare i diversi ceppi di virus e ricostruire la loro evoluzione (la cosiddetta analisi filogenomica) con livelli di precisione paragonabili ai metodi già in uso, ma in tempi estremamente più rapidi.
La caccia alle varianti è un procedimento molto complesso, che parte dai semplici tamponi. Il codice genetico dei campioni virali raccolti viene prima di tutto sequenziato con tecniche ad alta capacità (NGS, Next Generation Sequencing), poi deve essere tipizzato in modo da effettuare confronti tra i virus, trovare similitudini o differenze e, per queste ultime, stabilire le “distanze” genetiche tra un ceppo e l’altro. Un procedimento matematico il cui risultato è la classificazione delle varianti secondo la nomenclatura internazionale “PANGO”. Ma si può anche rendere graficamente: una serie di ramificazioni, quasi un cespuglio, che ci dicono la storia di una variante e, in qualche misura, come si sta diffondendo.
“I procedimenti comunemente in uso – dice Adriano Di Pasquale, Centro di Referenza Nazionale GENPAT - hanno il vantaggio di essere veloci, un elemento importante quando parliamo di una pandemia, ma non sono molto precisi. In altri termini, ci danno certamente una fotografia delle varianti e della loro evoluzione, ma è poco dettagliata. Aumentare la precisione, d’altro canto, richiede tempi molto lunghi, anche utilizzando computer potenti”.
L’algoritmo elaborato dai ricercatori IZSAM, invece, permette di aumentare notevolmente la risoluzione delle indagini genomiche mantenendo la rapidità necessaria. “Oltre ad individuare la diffusione di una variante – aggiunge Nicolas Radomski, GENPAT - ora possiamo seguirne la trasmissione, sia in termini di tempo che di spazio, osservando passo passo le ramificazioni che si stanno creando”.
Nato dall’analisi dei campioni di virus raccolti in Abruzzo e analizzati dallo Zooprofilattico di Teramo, il nuovo metodo matematico viene ora proposto a livello internazionale. “Pensiamo che possa essere un valido strumento per tutte le realtà che operano nel contesto della pandemia. – conclude Di Pasquale - È per questo che lo avevamo già messo a disposizione di tutti, sul portale Medrxiv. Abbiamo riscontrato un notevole interesse da parte di varie istituzioni, ed è già in corso una collaborazione con l’Istituto Nazionale di Sanità (INSA) portoghese”.

- Adriano Di Pasquale
Di Pasquale, A., Radomski, N., Mangone, I., Calistri, P., Lorusso, A., & Cammà, C. 2021. SARS-CoV-2 surveillance in Italy through phylogenomic inferences based on Hamming distances derived from pan-SNPs,-MNPs and-InDels. BMC genomics, 22(1).
Istituto Zooprofilattico Sperimentale
dell'Abruzzo e del Molise "G. Caporale"
Campo Boario | 64100 TERAMO | ITALIA
Telefono 0039.0861.3321 | Fax 0039.0861.332251
e-mail: archivioeprotocollo@izs.it
Posta elettronica certificata: protocollo@pec.izs.it
Partita IVA: 00060330677
Codice Fiscale: 80006470670





